全球首个非产毒艰难梭菌qPCR检测法问世,助力精准诊疗与抗生素管理
发布时间:2025-08-04 浏览次数:103 分享:
目前,艰难梭菌感染(CDI)诊断主要靠检测毒素蛋白(EIA)或毒素基因(NAAT),但都无法识别非产毒艰难梭菌(NTCD)。NTCD缺失PaLoc,常被75/115 bp非编码区替代,可能减少产毒菌株感染,降低复发性CDI,临床约30%“GDH-EIA+/毒素-EIA-”的不确定结果可能与NTCD有关,易致抗生素滥用,因此开发其特异性检测方法意义重大。
靶向NTCD特有区域检测方法开发
样本收集与处理
研究团队收集了2022-2023年休斯顿地区相关医院采用GDH-EIA两步法检测CDI的100份成年住院患者剩余粪便样本,-80℃储存,用DNeasy PowerSoil Pro Kit在QiaCube系统提取DNA,以70μL洗脱液洗脱保存。
PCR及qPCR方法开发
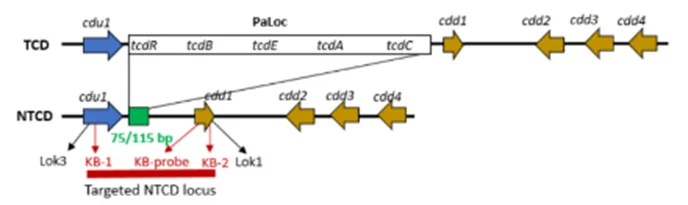
图 1 NTCD与产毒型艰难梭菌的PaLoc结构差异及检测靶点示意图
设计针对NTCD特有115bp非编码区的KB-1/KB-2引物(约522bp),与已发表的Lok3/lok1引物对比,经Sanger测序和BLAST验证特异性。
构建的NTCD qPCR体系采用上述引物及FAM标记探针,在QuantStudio5系统反应,20μL体系含25ng DNA模板及特定浓度引物、探针,反应条件为95℃预变性10分钟,随后40个循环的95℃15秒和60℃1分钟。
敏感性与特异性验证
使用NTCD参考菌株、产毒艰难梭菌及相关梭菌的基因组DNA,经10倍梯度稀释确定qPCR分析敏感性,检测不同菌株评估特异性,同时通过含不同浓度菌株的粪便spike实验验证直接检测效果。
临床样本检测
用新开发的qPCR检测95份经GDH-EIA两步法检测的临床粪便样本中的NTCD,统计不同GDH-EIA结果组的NTCD阳性比例及DNA含量。
关键结果:高特异性与临床应用价值凸显
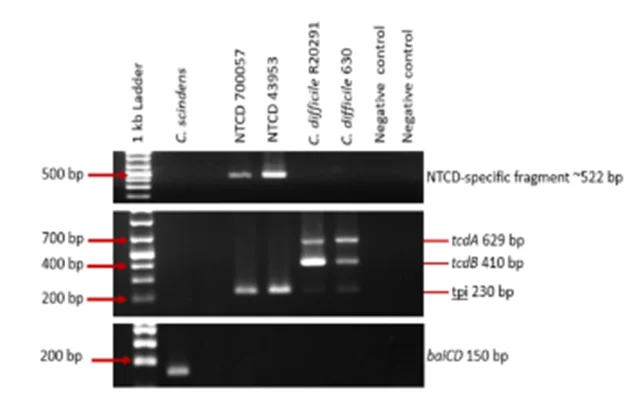
图 2 NTCD特异性PCR在参考菌株中的特异性验证结果
如图1,研究团队设计了针对NTCD特有非编码区域的KB-1/KB-2引物和FAM标记探针,通过优化qPCR反应条件,建立了可定量检测NTCD的方法。该方法经参考菌株验证,特异性极强——仅扩增NTCD,对产毒艰难梭菌及其他相关菌株无交叉反应。
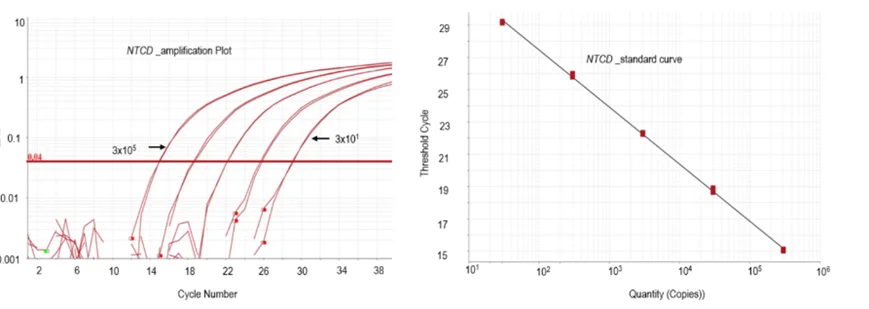
图 3 NTCD qPCR标准曲线与扩增效率分析图
NTCD qPCR在3×10至3×10⁶ gDNA范围内线性良好(R²=0.999,P<0.0001),对产毒菌株检测阴性,能检出NTCD菌株及产毒与NTCD混合菌株中的NTCD。
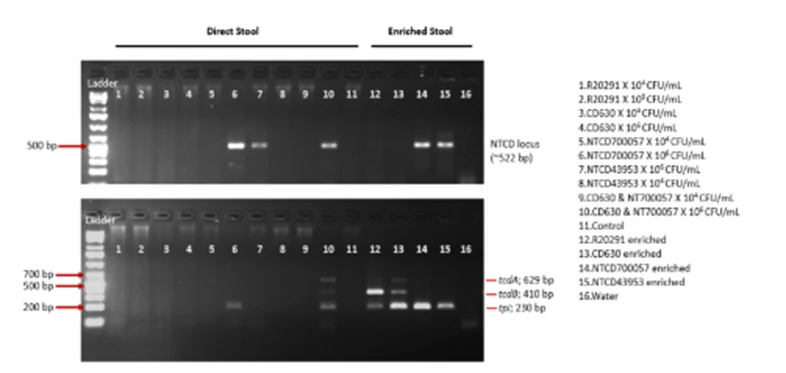
图 4 粪便样本富集与否对NTCD PCR检测效果的影响
如图4,在95份临床样本中,GDH-EIA-/-的25份未检出NTCD;GDH-EIA+/-的25份中,20%(5份)检出,median NTCD DNA为33,039(IQR:22,449–45,688);GDH-EIA+/+的23份中,8.7%(2份)检出,median为370(IQR:159–583),总体住院患者NTCD定植率7%。
该研究表明,新qPCR方法可高效识别NTCD,有助于解释CDI诊断中的不确定结果,减少不必要的抗生素使用,为CDI的精准防控提供了新思路。
研究亮点
1. 开发出针对NTCD的qPCR方法,通过KB-1/KB-2引物和探针精准识别NTCD,填补现有诊断无法检测NTCD的空白。
2. 新方法仅扩增NTCD相关区域,对产毒菌株及其他相关菌株无交叉反应;在3×10至3×10⁶ gDNA范围内线性良好(R²=0.999,P<0.0001),分析敏感性高。
3. 明确NTCD在不同GDH-EIA结果组的定植情况,为解释CDI诊断不确定结果、避免不必要抗生素使用提供依据,总体住院患者NTCD定植率为7%。
4. KB-1/KB-2引物相比Lok3/lok1引物能扩增更多NTCD菌株,检测效果更优。
原文链接:https://doi.org/10.1128/spectrum.01608-24
来源:微生物安全与健康网,作者~肖锦琦。