机器学习辅助的代谢标记传感阵列用于快速识别病原菌
发布时间:2024-08-30 浏览次数:481
快速鉴定病原菌是确保适当给药以减轻抗生素耐药性进展的关键先决条件。化学鼻策略通过使用化学传感阵列和机器学习算法模拟动物嗅觉,为物种识别和区分提供了一种潜在的方法。其中,荧光传感器阵列因其高灵敏度而备受关注。而当前的大多数荧光传感阵列策略依赖于探针和病原菌之间的非特异性相互作用。这些相互作用很容易被周围环境的变化所破坏,导致它们在复杂基质中的可行性大大降低。同时,这些方法难以应用于复杂的样本矩阵和识别训练集之外的未知物种。
代谢标记是一种不干扰其生理过程的情况下将化学基团纳入生物体的有效方法。其中,外源性D-氨基酸在细菌特异性识别方面具有独特的优势。通常,外源性D-氨基酸通过代谢过程整合,决定外源D-氨基酸标记的有三种酶,即D,D-转肽酶,D,D-羧基肽酶,L,D-转肽酶。外源性D-氨基酸可以通过D,D-转肽酶或L,D-转肽酶介导的代谢方式整合进细菌肽聚糖结构。通过代谢途径整合的D-氨基酸可能会被D,D-羧肽酶进一步水解,降低外源性D-氨基酸融入肽聚糖结构的总体水平。其中D,D-转肽酶和D,D-羧基肽酶存在于所有细菌中,但其类型和数量各不相同。而L,D-转肽酶仅存在于有限种类的细菌中。这种差异导致不同菌种间外源D-氨基酸整合程度不同。由于这些酶的活性是动态变化的,与细菌的生长和分裂密切相关,因此这些酶介导的代谢标记可能在不同条件下表现出差异,为病原菌的鉴定提供了有价值的信息。
本研究基于肽聚糖代谢过程中酶活性动态变化,建立了一种用于区分病原菌的代谢标记化学鼻方法(图1)。对数生长期的细菌在pH=5和7的条件下被3-叠氮基-D-丙氨酸(D-Ala-N3)代谢标记。然后在不同时间点用二苯并环辛炔功能化的上转换纳米颗粒(DBCO-UCNPs)点击以产生一系列荧光信号输出。输出的信号通过机器学习算法处理,以识别和区分病原菌。这种感知策略可以精确区分不同的菌株和物种。由于反应变化与病原菌的内在特性,即代谢活性有关,因此,该策略对复杂基质具有高耐受性,并能够精确识别训练集之外的物种。其可靠、准确的病原菌鉴定能力使其在临床检测和诊断中具有广阔的应用前景。
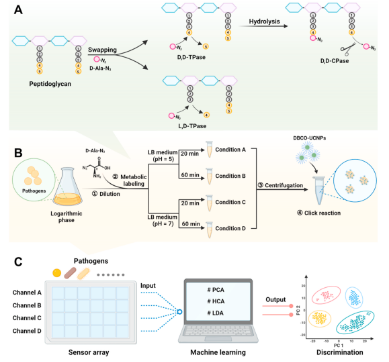
图1 化学鼻策略鉴别病原菌的示意图。(A)D, D-TPase和L, D-TPase分别介导外源性D-Ala-N3交换五肽链的第5和第4个D-氨基酸;D,D-羧基肽酶会水解五肽末端的D-氨基酸,包括外源的D-氨基酸。(B)在4种条件下,用D-Ala-N3对病原菌进行代谢标记,然后通过点击反应进行信号输出。(C)借助机器学习识别病原菌的传感器阵列。
研究首先通过热解方法合成上转换纳米粒子,然后通过配体交换机理,使用二苯并环辛炔(DBCO)将上转换纳米颗粒(UCNPs)功能化,形成DBCO-UCNPs纳米颗粒。相比之下,DBCO-UCNPs具有更高的荧光强度,在980 nm激光激发下,观察到521、541和654 nm处的荧光发射,这与未修饰的上转换纳米粒子相同。由于相对较高的灵敏度,选择541 nm处收集荧光发射,用于后续检测。
然后研究了传感策略的可行性。在不同培养条件下不同代谢标记菌株在发生点击后的荧光强度变化,确认是否可以作为识别元素用于多种病原菌的鉴别。并在不同培养条件下连续测定3天,评估策略的稳定性。
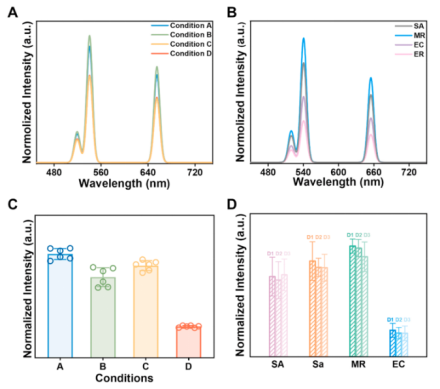
图2 传感策略的可行性分析。(A)不同培养条件下代谢标记的金黄色葡萄球菌(ATCC25923(Sa))的荧光光谱。(B)不同模型菌株(金黄色葡萄球菌 ATCC 29213(SA)、金黄色葡萄球菌 ATCC 43300(MR)、大肠埃希氏菌O157:H7(EC)和大肠埃希氏菌ER2738)在pH=7条件下代谢标记60 min并与DBCO修饰的上转换纳米粒子发生点击反应。(C)不同条件下代谢标记的ER与DBCO修饰的上转换纳米粒子点击反应之后的归一化荧光强度;(D)在条件B下连续测试3天内策略的稳定性。条件A:pH=5,代谢标记20 min;条件B:pH=7,代谢标记20 min;条件C:pH=5,代谢标记60 min;条件D:pH=7,代谢标记60 min。
接着以8种菌株(5株革兰氏阳性菌和3株革兰氏阴性菌,包括革兰氏阳性和阴性抗生素敏感菌株和耐药菌株)作为模型菌株进行检测,评估该方法在训练集内的鉴别能力。
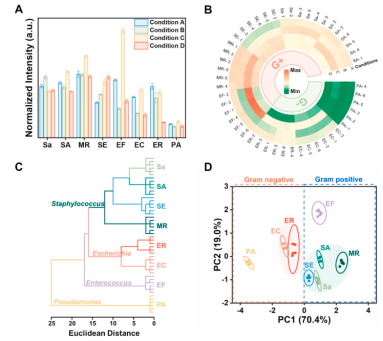
图3 训练集中8种细菌的鉴别。8种菌株的(A)荧光指纹图谱;(B)热图;(C)聚类数突图;(D)PCA聚类图。
总之,本研究提供了一种基于肽聚糖代谢过程中酶活性动态变化的化学鼻策略,用于快速准确地识别病原菌。借助上转换纳米颗粒将不同代谢标记条件下细菌酶活性的变化转化为荧光信号进行输出,用于病原菌鉴定。所提出的策略在复杂的基质中具有高耐受性,如尿液。此外,革兰氏阴性菌和革兰氏阳性菌之间不同的代谢标记行为有助于根据革兰氏特性鉴别病原菌。进一步将抗生素依赖传感通道纳入所建立的传感策略有利于在抗生素药敏测试中区分抗生素耐药性。
论文链接:https://doi.org/10.1016/j.bios.2024.116264
来源:微生物安全与健康网,作者~冯燕梅





